20 Análise de sobrevida
Os conteúdos das seções 20.1, 20.2 e 20.3 podem ser visualizados neste vídeo.
20.1 Introdução
A análise de sobrevida visa a fornecer estimativas da probabilidade de um indivíduo experimentar um dado evento de interesse e eventualmente comparar curvas de sobrevida entre diferentes estratos de pacientes. O nome “análise de sobrevida” deriva da utilização desse tipo de análise quando o evento de interesse é a ocorrência ou não de morte, mas ela pode ser utilizada para outros eventos, como recidiva de câncer, infecção, etc.
Essa é uma área bastante complexa e há inúmeras publicações dedicadas ao assunto. Este capítulo mostra como construir curvas de sobrevida no R para dados com censura à direita, como estimar probabilidades de sobrevida por meio do método de Kaplan-Meier e como comparar curvas de sobrevida por meio do teste log-rank.
20.2 Conjunto de dados utilizado neste capítulo
Vamos utilizar o conjunto de dados cancer do pacote survival (LGPL-2 | LGPL-2.1 | LGPL-3).
Essa base contém dados relativos à sobrevida de 228 indivíduos com câncer avançado do pulmão do North Central Cancer Treatment Group. Neste capítulo, será utilizado o plugin RcmdrPlugin.survival. Ao instalar esse plugin com as dependências, o pacote survival também será instalado.
Os passos para a instalação desse pacote são os mesmos utilizados para a instalação do R Commander, seção A.6.
Os comandos abaixo carregam o plugin RcmdrPlugin.survival e o conjunto de dados cancer no R Commander. Após a execução desses comandos, selecione o conjunto de dados cancer como conjunto de dados ativo.
library(RcmdrPlugin.survival)
data(cancer, package="survival")A figura 20.1 mostra a estrutura desse conjunto de dados.

Figura 20.1: Estrutura do conjunto de dados cancer do pacote survival.
O comando abaixo lista os cinco primeiros registros desse conjunto de dados.
head(cancer, 5)## inst time status age sex ph.ecog ph.karno pat.karno meal.cal wt.loss
## 1 3 306 2 74 1 1 90 100 1175 NA
## 2 3 455 2 68 1 0 90 90 1225 15
## 3 3 1010 1 56 1 0 90 90 NA 15
## 4 5 210 2 57 1 1 90 60 1150 11
## 5 1 883 2 60 1 0 100 90 NA 0Para construirmos uma curva de sobrevida para os pacientes com câncer de pulmão, precisamos trabalhar com as seguintes variáveis:
time: tempo de sobrevida em dias
status: status da censura (1=censura, 2=morte)
Em geral, em estudos em que se busca identificar se um indivíduo experimentou um determinado evento (desfecho) de interesse durante a duração do estudo e o tempo entre a entrada no estudo e a ocorrência do evento, é preciso incluir uma variável que indica se o indivíduo experimentou ou não o evento durante o período em que ele(a) participou do estudo. Caso o indivíduo não tenha experimentado o desfecho no período em que ele(a) foi observado, dizemos que houve uma censura, e o indivíduo foi censurado.
Um sujeito pode ser censurado devido às seguintes razões:
- perda de acompanhamento;
- retirada do estudo;
- não ocorreu o evento de interesse ao final do período de estudo.
Essas três razões se aplicam a censuras conhecidas como censuras à direita, porque ocorrem após o paciente entrar no estudo. Censuras à esquerda e censuras de intervalo também são possíveis, mas este texto irá tratar somente de censuras à direita.
No conjunto de dados cancer, a variável status indica se ocorreu ou não uma “censura” para o respectivo paciente, sendo o evento de interesse a morte. O status igual a 1 significa que houve censura, ou seja, até o momento em que o paciente foi acompanhado no estudo, ele(a) ainda estava vivo(a) e o valor do tempo de sobrevida corresponde ao tempo decorrido desde a entrada do paciente no estudo até o momento em que ele(a) foi retirado(a) do estudo. O tempo de sobrevida desse paciente é maior do que o indicado no estudo, mas não sabemos o que aconteceu com ele(a) após o momento de saída do estudo. O status igual a 2 indica que o paciente morreu durante o acompanhamento no estudo e o valor do tempo de sobrevida corresponde ao tempo desde a entrada do paciente no estudo até a sua morte.
Assim, se observarmos os três primeiros registros do conjunto de dados cancer, iremos verificar que os dois primeiros pacientes morreram nos dias 306 e 455 após o início do acompanhamento de cada um deles, respectivamente. Nesses dois casos, não houve censura (status = 2). O terceiro paciente foi censurado (status = 1) após 1010 dias de acompanhamento. Assim a sobrevida do terceiro paciente é superior a 1010 dias, mas não sabemos exatamente qual o seu valor.
Observação: apesar de o conjunto de dados cancer codificar os pacientes que foram censurados com o valor 1 e os que morreram com o valor 2, em geral, são utilizados os valores 0 para indicar a censura e o valor 1 para indicar os indivíduos que experimentaram o evento estudado.
20.3 Obtendo a curva de sobrevida no R
A curva de sobrevida mostra, para cada instante de tempo, a probabilidade de um indivíduo sobreviver além daquele instante de tempo.
Nesta seção, vamos construir uma curva de sobrevida para os pacientes do conjunto de dados cancer a partir do R Commander. A seção seguinte irá explicar como essa curva é obtida.
Uma vez carregados o plugin RcmdrPlugin.survival e tendo o conjunto de dados cancer como conjunto ativo, para gerar a curva de sobrevida, selecionamos a seguinte opção do menu do R Commander:
\[\text{Estatísticas} \Rightarrow \text{Análise de Sobrevida} \Rightarrow \text{Função estimada de sobrevida}\]
Na tela Função de Sobrevida (figura 20.2), selecionamos na primeira lista a variável que indica o tempo até a ocorrência do evento ou censura (time no conjunto de dados cancer). Na segunda lista, selecionamos a variável que indica se o indivíduo foi censurado ou não (nesse exemplo, a variável é status). Em tipo de censura, vamos selecionar a opção Right.
Figura 20.2: Tela para especificar as variáveis que serão utilizadas para construir a curva de sobrevida.
Na aba Opções (figura 20.3), há diversas opções que podem ser configuradas para construir a curva de sobrevida. Vamos manter as opções padrão:
- intervalos de confiança: logaritmo;
- gráfico com intervalos de confiança: default, correspondente à opção sim;
- nível de confiança : 95%;
- método: Kaplan-Meier;
- método de variância: Greenwood;
- quantis para estimar: P25, Mediana, P75;
- indicar os tempos de censura: sim;
- resumo: padrão.
Figura 20.3: Tela para definir as opções para construir a curva de sobrevida.
Ao clicarmos em OK, a seguinte sequência de comandos é executada:
- A função survfit gera uma série de componentes utilizados para construir a curva de sobrevida. Esses componentes são armazenados no objeto .Survfit. Um resumo do objeto .Survfit é exibido na tela, indicando a função chamada, o número de pessoas observadas (n), o número de mortes (events), a mediana do tempo de sobrevida (median), que é o tempo no qual a probabilidade de sobrevida é de 50%, e os limites inferior (0.95LCL) e superior (0.95UCL) do intervalo de confiança para a mediana do tempo de sobrevida.
.Survfit <- survfit(Surv(time, status, type="right") ~ 1, conf.type="log",
conf.int=0.95, type="kaplan-meier",
error="greenwood", data=cancer)
.Survfit## Call: survfit(formula = Surv(time, status, type = "right") ~ 1, data = cancer,
## error = "greenwood", conf.type = "log", conf.int = 0.95,
## type = "kaplan-meier")
##
## n events median 0.95LCL 0.95UCL
## [1,] 228 165 310 285 363- A curva de sobrevida é exibida por meio da função plot, aplicada sobre o objeto .Survfit (figura 20.4). Ela mostra, como dito antes, para cada instante de tempo, a probabilidade de um indivíduo sobreviver além daquele instante de tempo. Essa curva é decrescente com o tempo.
par(mar=c(3,3,2,1))
plot(.Survfit, mark.time=TRUE)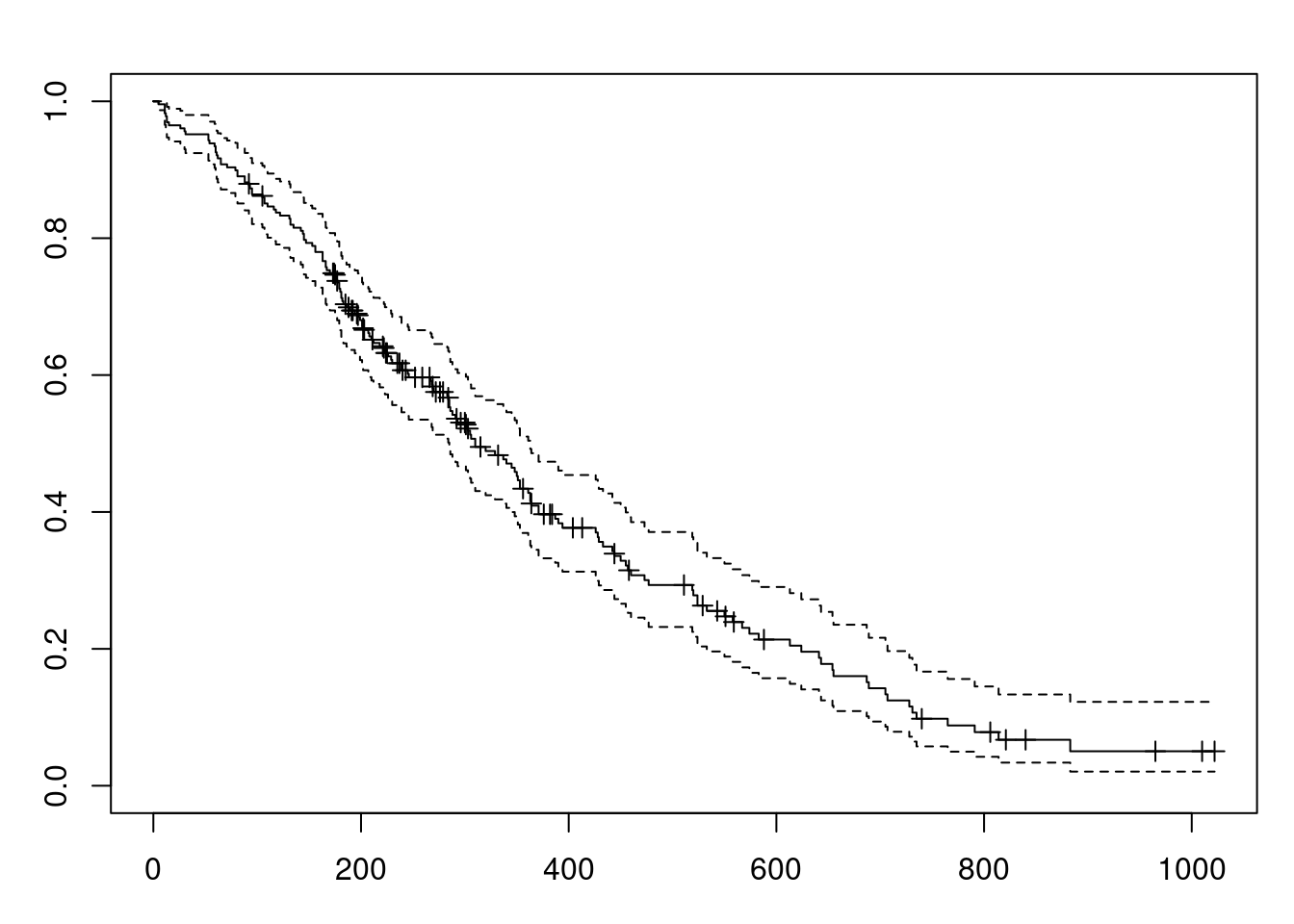
Figura 20.4: Curva de sobrevida pelo método de Kaplan-Meier para o conjunto de dados cancer.
- São exibidos os valores das estimativas e respectivos intervalos de confiança para o P25, a mediana e o P75 do tempo de sobrevida por meio da função quantile.
quantile(.Survfit, quantiles=c(.25,.5,.75))## $quantile
## 25 50 75
## 170 310 550
##
## $lower
## 25 50 75
## 145 285 460
##
## $upper
## 25 50 75
## 197 363 654- O objeto .Survfit é removido da área de trabalho.
remove(.Survfit)20.4 Estimando a probabilidade de sobrevida
Os conteúdos desta seção e da seção seguinte (seção 20.5) podem ser visualizados neste vídeo.
O método de Kaplan-Meier é um método não paramétrico, sendo o mais utilizado para estimar tempos de sobrevida e probabilidades de sobrevida. Ele resulta em uma função degrau, onde há um degrau para baixo a cada instante em que um evento ocorre.
A probabilidade de sobrevida em um certo instante de tempo é a probabilidade de um indivíduo experimentar o evento de interesse após o instante t.
A função Surv do pacote survival gera um valor para cada indivíduo que é o seu tempo de sobrevida, seguido por um sinal de + se o indivíduo foi censurado. Vamos ver os primeiros 10 valores:
Surv(cancer$time, cancer$status)[1:10]## [1] 306 455 1010+ 210 883 1022+ 310 361 218 166Observamos que esses valores correspondem ao tempo de sobrevida dos 10 primeiros indivíduos do conjunto de dados cancer, indicando que os indivíduos 3 e 6 foram censurados (status = 1).
Para estimar as probabilidades de sobrevida, os tempos de sobrevida são ordenados em ordem crescente. A função a seguir mostra os 15 menores tempos de sobrevida do conjunto de dados cancer, com a censura indicada pelo sinal + após o valor do tempo. Podemos ver que um indivíduo sobreviveu somente 5 dias, três indivíduos sobreviveram 11 dias, um indivíduo sobreviveu 12 dias, dois indivíduos sobreviveram 13 dias e assim por diante.
sort(Surv(cancer$time, cancer$status))[1:15]## [1] 5 11 11 11 12 13 13 15 26 30 31 53 53 54 59Os valores gerados pela função Surv são utilizados pela função survfit para gerar o objeto .Survfit na seção anterior. Vamos gerar novamente esse objeto e compreender como são estimadas as probabilidades de sobrevida após um certo tempo t e como é construída a curva de sobrevida.
O objeto .Survfit possui diversos componentes, cujos nomes podem ser obtidos por meio da função names aplicada a .Survfit, que nos permite construir uma tabela que gera as probabilidades de sobrevida de pacientes com câncer de pulmão em cada instante de tempo.
.Survfit <- survfit(Surv(time, status, type="right") ~ 1, conf.type="log",
conf.int=0.95, type="kaplan-meier",
error="greenwood", data=cancer)
names(.Survfit)[1:6]## [1] "n" "time" "n.risk" "n.event" "n.censor" "surv"names(.Survfit)[7:12]## [1] "std.err" "cumhaz" "std.chaz" "type" "logse" "conf.int"names(.Survfit)[13:16]## [1] "conf.type" "lower" "upper" "call"Utilizando os componentes time, n.risk, n.event, n.censor e surv de .Survfit, podemos montar uma tabela que dá a estimativa da probabilidade de sobrevida até pelo menos cada um dos tempos de sobrevida dos pacientes do conjunto de dados cancer. O comando abaixo mostra as 10 primeiras linhas desta tabela.
head(with(.Survfit, data.frame(time, n.risk, n.event, n.censor, surv)), 10)## time n.risk n.event n.censor surv
## 1 5 228 1 0 0.9956140
## 2 11 227 3 0 0.9824561
## 3 12 224 1 0 0.9780702
## 4 13 223 2 0 0.9692982
## 5 15 221 1 0 0.9649123
## 6 26 220 1 0 0.9605263
## 7 30 219 1 0 0.9561404
## 8 31 218 1 0 0.9517544
## 9 53 217 2 0 0.9429825
## 10 54 215 1 0 0.9385965Para cada linha da tabela acima, time indica o tempo de sobrevida, n.risk o número de pacientes em risco de experimentar o evento antes do valor indicado por time, n.event indica o número de indivíduos que experimentaram o evento no tempo mostrado na linha corrente, n.censor indica o número de indivíduos que foram censurados no tempo mostrado na linha corrente, surv é a probabilidade de sobrevida até pelo menos o instante indicado pela linha corrente.
Assim a primeira linha indica que 1 paciente morreu no \(5^o\) dia e nenhum foi censurado nesse dia. 228 pacientes entraram no estudo. Logo 227 pacientes sobreviveram até pelo menos o quinto dia de acompanhamento. Então a probabilidade de um paciente sobreviver ao instante 5 é estimada pelo número de pacientes que sobreviveram ao \(5^o\) dia de acompanhamento dividido pelo número de pacientes em risco de morte no início do acompanhamento (227/228 = 0,9956).
A segunda linha indica que 3 pacientes morreram no \(11^o\) dia e nenhum foi censurado nesse dia. 227 pacientes estavam em risco de morrer após o \(5^o\) dia. Logo 224 pacientes sobreviveram ao \(11^o\) quinto dia de acompanhamento. Então a probabilidade de um paciente sobreviver ao \(11^o\) dia, tendo sobrevivido ao \(5^o\) dia, é estimada pelo número de pacientes que sobreviveram ao \(11^o\) dia de acompanhamento dividido pelo número de pacientes em risco do evento após o \(5^o\) dia (224/227 = 0,9868). Finalmente a probabilidade de o paciente sobreviver a 11 dias é igual ao produto da probabilidade de ele sobreviver a 5 dias (0,9956) pela probabilidade de ele sobreviver a 11 dias, tendo sobrevivido a 5 dias (0,9868). Logo a probabilidade de um indivíduo sobreviver a 11 dias é 0,9825 (0,9956 x 0,9868).
A figura 20.5 mostra o passo a passo para os cálculos da probabilidade de sobrevida em cada instante pelo método de Kaplan-Meier. As colunas n.risk, tempo, n.event e n.censor possuem o mesmo significado dos componentes n.risk, time, n.event e n.censor do objeto .Survfit.
A coluna probabilidade de sobrevida no período ti-1 a ti mostra a probabilidade de um indivíduo sobreviver ao instante ti, caso ele esteja vivo no instante ti-1, p(ti) = P(t>ti| t>ti-1), e é calculada, em cada linha, pela expressão:
\[p(t_i) = P(t>t_i\ |\ t>t_{i-1}) = \frac{n.risk_i - n.event_i}{n.risk_i}\]
O número de pacientes em risco na linha i é calculado subtraindo do número de pacientes em risco na linha anterior (i-1) a soma do número de pacientes que morreram com o número de pacientes censurados, ambos na linha (i-1):
\[n.risk_i = n.risk_{i-1} - n.event_{i-1} - n.censor_{i-1}\]
O número de pacientes em risco na linha i é calculado subtraindo do número de pacientes em risco na linha A coluna probabilidade de sobrevida no instante ti mostra a probabilidade de um indivíduo sobreviver ao instante ti, P(ti)=P(t>ti), e é calculada, na linha i, pelo produto da probabilidade de sobrevida no instante ti-1 (linha anterior) pela probabilidade de sobrevida no período ti-1 a ti na mesma linha.
\[P(t_i) = P(t>t_i) = P(t_{i-1}).p(t_i)\]
A linha destacada pelo retângulo vermelho na figura 20.5 mostra esse cálculo para a probabilidade de sobrevida no instante t2 = 11 (0,9825), que é igual ao produto da probabilidade de sobrevida no instante t1 = 5 (0,9956) pela probabilidade de sobrevida no período t1 a t2 (0,9868). Seguindo esse raciocínio, calcula-se as probabilidades de sobrevida nos demais instantes.
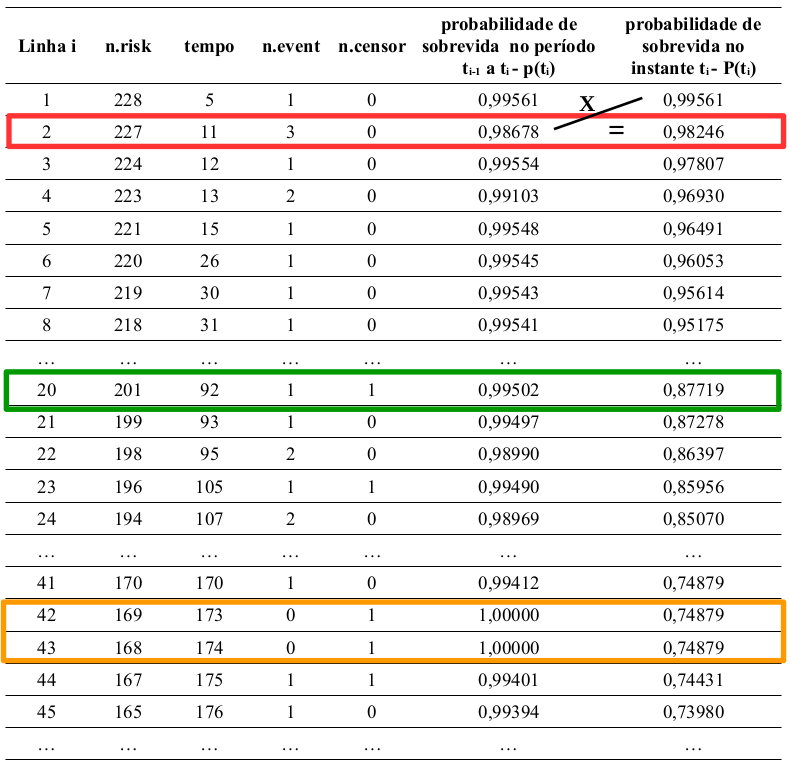
Figura 20.5: Tabela para obter as estimativas da probabilidade de sobrevida pelo método de Kaplan-Meier.
O retângulo verde na figura 20.5 mostra uma situação onde ocorre uma censura. Os pacientes censurados em um instante de tempo causam uma redução no número de pacientes sob risco do evento nos instantes posteriores.
O retângulo alaranjado na figura 20.5 mostra duas situações onde ocorre uma censura no instante t42 = 173 e outra censura no instante t43 = 174, mas não ocorreu nenhuma morte entre o instante t41 = 170 e t43 = 174. Podemos observar que as probabilidades de sobrevida nos instantes t42 e t43 continuam iguais à probabilidade de sobrevida no instante t41. Portanto precisamos atualizar as estimativas das probabilidades de sobrevida somente nos instantes em que ocorrem o evento de interesse.
A partir da tabela da figura 20.5, a curva de sobrevida é construída, plotando para cada instante em que ocorreu o evento de interesse a probabilidade de sobrevida nesse instante. Os pontos assim obtidos são unidos por uma função em degrau, da seguinte forma (figura 20.6): dados dois pontos (ti-1, P[ti-1]) e (ti, P[ti]), eles são unidos por uma linha horizontal que vai de (ti-1, P[ti-1]) a (ti, P[ti-1]) e, em seguida, por uma linha vertical que vai de (ti, P[ti-1]) a (ti, P[ti]).
![Como os pontos de coordenadas (ti-1, P[ti-1]) e (ti, P[ti]) são unidos para construir a curva de sobrevida pelo método de Kaplan-Meier.](imagens/analiseSobrevida/ligacaoProbSobrevida.png)
Figura 20.6: Como os pontos de coordenadas (ti-1, P[ti-1]) e (ti, P[ti]) são unidos para construir a curva de sobrevida pelo método de Kaplan-Meier.
20.5 Obtendo as probabilidades de sobrevida em instantes específicos
Uma quantidade de interesse na análise de sobrevida é a probabilidade de sobreviver além de um certo instante de tempo. O comando abaixo mostra como estimar a probabilidade de sobreviver além de 1 e 2 anos para o conjunto de dados cancer. No argumento times, especificamos um vetor com dois valores (365,25 e 730,5), que correspondem, respectivamente, a 1 e a 2 anos, já que estamos medindo o tempo em dias e, em média, um ano contém 365,25 dias.
summary(survfit(Surv(time, status) ~ 1, data=cancer), times=c(365.25, 730.5))## Call: survfit(formula = Surv(time, status) ~ 1, data = cancer)
##
## time n.risk n.event survival std.err lower 95% CI upper 95% CI
## 365 65 121 0.409 0.0358 0.3447 0.486
## 730 13 38 0.116 0.0283 0.0716 0.187Nos resultados acima, a coluna survival fornece as probabilidades de sobrevida, que são iguais a 0,41 e 0,12 para um e dois anos, respectivamente. As colunas std.err, lower 95% CI e upper 95% CI fornecem os valores do erro padrão, o limite inferior do intervalo de confiança e o limite superior do intervalo de confiança para a probabilidade de sobrevida além do primeiro e segundo ano, respectivamente.
Nesse exemplo, o erro padrão da probabilidade de sobrevida além de um certo instante ti, EP[P(ti)], é calculado de acordo com a proposta de Greenwood (Greenwood 1926):
\[EP[P(t_i)] = P(t_i). \sqrt{\sum_{j=1}^{i}{\frac{n.event_j}{n.risk_j - n.event_j}}}\]
Os limites inferior e superior para o intervalo com nível de confiança \((100 - \alpha)\%\) para a probabilidade de sobrevida além de um certo instante ti são dados por:
\[[max(P(t_i) - z_{1-\alpha/2} . EP[P(t_i)], 0), min(P(t_i) + z_{1-\alpha/2} . EP[P(t_i)], 1)]\]
ou seja, o limite inferior do intervalo de confiança para P(ti) é o máximo entre 0 e \(P(t_i)-z_{1-\alpha/2} . EP[P(t_i)]\) e o limite superior do intervalo de confiança para P(ti) é o mínimo entre 1 e \(P(t_i) + z_{1-\alpha/2} . EP[P(t_i)]\).
A fórmula de Greenwood é acurada somente assintoticamente, ou seja, para valores suficientemente grandes para n.riski. Há diversas outras propostas que visam a tornar mais acuradas as estimativas para amostras não muito grandes.
20.6 Obtendo a curva de sobrevida para diferentes estratos
Os conteúdos desta seção e da seção seguinte (seção 20.7) podem ser visualizados neste vídeo.
Frequentemente, deseja-se comparar curvas de sobrevida para diferentes tratamentos para uma determinada condição clínica, ou comparar curvas de sobrevida para diferentes estratos de pacientes. Vamos ilustrar como construir curvas de sobrevida para diferentes níveis de uma variável categórica, usando novamente o conjunto de dados cancer, o qual possui a variável categórica sex.
Como a variável sex está como um vetor numérico, vamos convertê-la para fator por meio do comando:
cancer <- within(cancer, {
sex <- factor(sex, labels = c('masculino', 'feminino'))
})Para construir curvas de sobrevida para diferentes níveis de uma variável categórica, escolhemos novamente a opção:
\[\text{Estatísticas} \Rightarrow \text{Análise de Sobrevida} \Rightarrow \text{Função estimada de sobrevida}\]
Na aba Dados (figura 20.7), selecionamos na primeira lista a variável time e na segunda lista, a variável status). Vamos selecionar a opção Right em tipo de censura e a variável sex no item estrato.
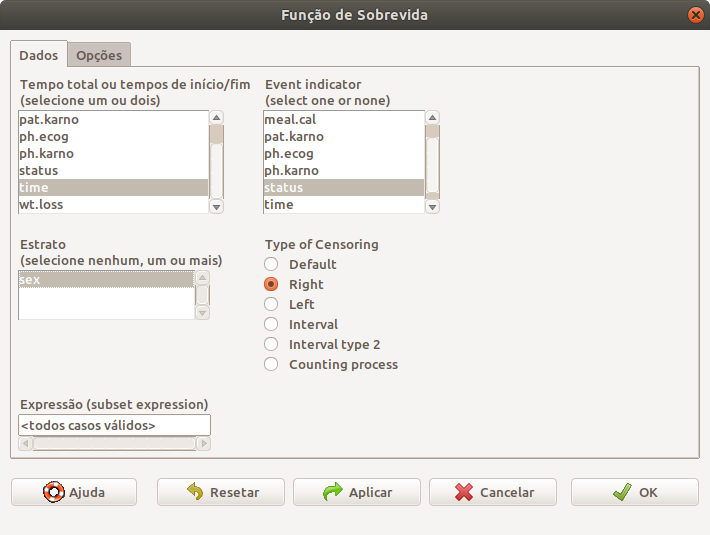
Figura 20.7: Tela para configurar as variáveis que serão utilizadas para construir a curva de sobrevida para diferentes estratos.
Ao clicamos em OK, os comandos a seguir serão executados, seguidos dos resultados e das curvas de sobrevida para cada estrato (figura 20.8).
.Survfit <- survfit(Surv(time, status, type="right") ~ sex, conf.type="log",
conf.int=0.95, type="kaplan-meier",
error="greenwood", data=cancer)
.Survfit## Call: survfit(formula = Surv(time, status, type = "right") ~ sex, data = cancer,
## error = "greenwood", conf.type = "log", conf.int = 0.95,
## type = "kaplan-meier")
##
## n events median 0.95LCL 0.95UCL
## sex=masculino 138 112 270 212 310
## sex=feminino 90 53 426 348 550plot(.Survfit, col=1:2, lty=1:2, mark.time=TRUE)
legend("bottomleft", legend=c("sex=masculino","sex=feminino"), col=1:2,
lty=1:2, bty="n")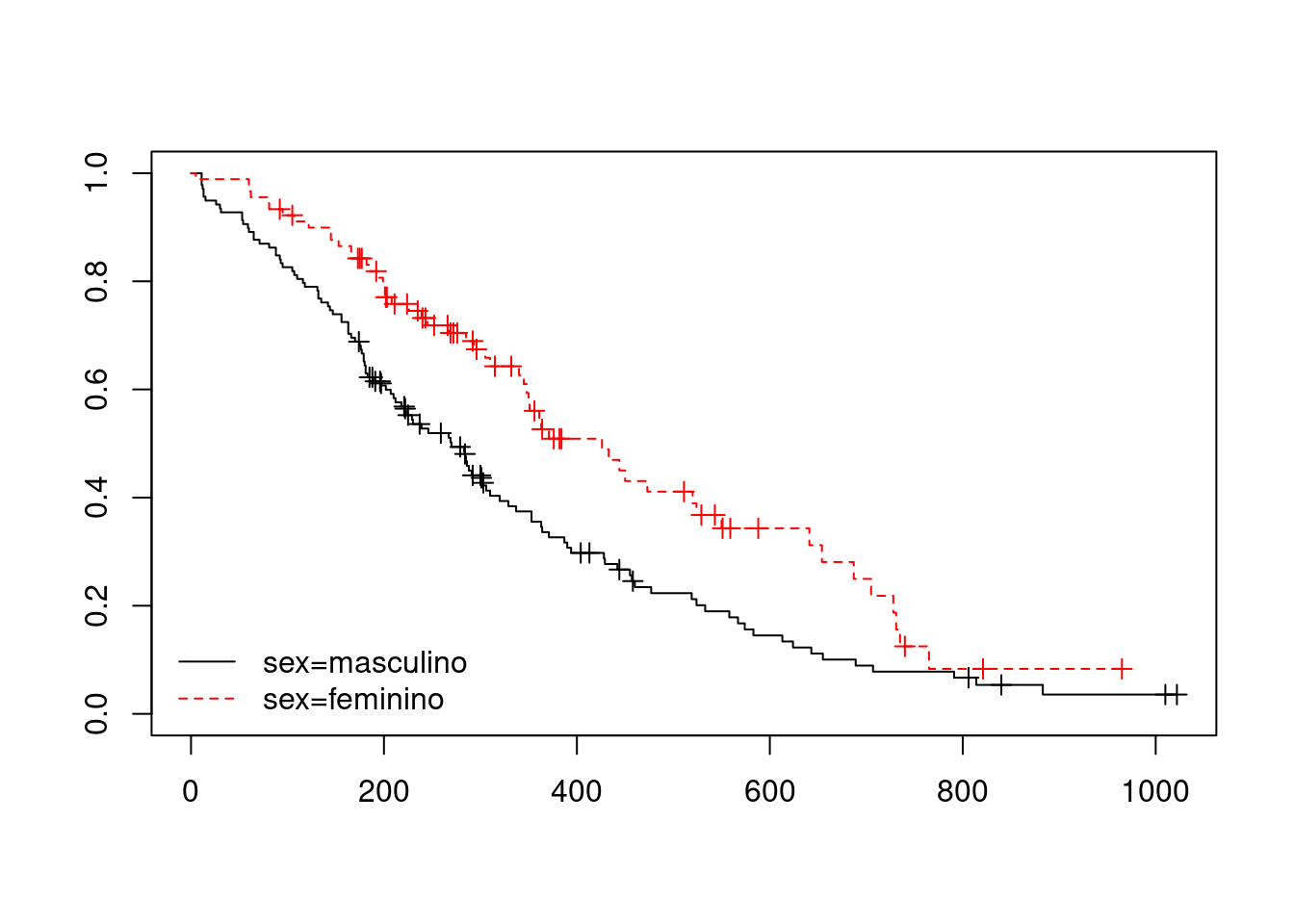
Figura 20.8: Curvas de sobrevida para cada sexo no conjunto de dados cancer.
quantile(.Survfit, quantiles=c(.25,.5,.75))## $quantile
## 25 50 75
## sex=masculino 144 270 457
## sex=feminino 226 426 687
##
## $lower
## 25 50 75
## sex=masculino 107 212 387
## sex=feminino 186 348 550
##
## $upper
## 25 50 75
## sex=masculino 177 310 574
## sex=feminino 340 550 NAremove(.Survfit)Podemos ver que a sobrevida é maior entre as mulheres do que nos homens, sendo a mediana do tempo de sobrevida igual a 270 e 426 dias para os homens e as mulheres, respectivamente.
20.7 Comparação de funções de sobrevida em diferentes estratos
Existem vários testes estatísticos que avaliam a hipótese nula de que não há diferença entre as funções de sobrevida entre diferente estratos. Um método frequentemente usado é o teste log-rank. Diversas formas desta estatística foram publicadas por diferentes estatísticos, por isso esse teste é conhecido por diversos nomes. Esse teste compara o número de eventos observados em cada grupo com o número de eventos esperados se os dois grupos são combinados em um só. Vamos ilustrar uma das formas como esse teste é realizado na função survdiff do pacote survival, conhecida como teste log-rank de Mantel-Haenszel.
Para realizar o teste log-rank de Mantel-Haenszel, uma tabela como a tabela 20.1 é construída. Nessa tabela, vamos supor que temos dois estratos ou grupos, onde A representa um estrato e B o outro. Cada linha i da tabela contém os tempos de ocorrência do evento de interesse em ordem crescente (ti), o número de indivíduos em risco em cada estrato (nAi e nBi), o número total de indivíduos em risco (nTi), o número de eventos observados no tempo ti em cada estrato (nAoi e nBoi) e nos dois estratos em conjunto (nABoi = nAoi + nBoi), e o número de eventos esperados de ocorrer em cada estrato no tempo ti (nAei e nBei).
| tempo | Grupo A | Grupo B | Total | Grupo A | Grupo B | Total | Grupo A | Grupo B |
|---|---|---|---|---|---|---|---|---|
| t1 | nA1 | nB1 | nT1 | nAo1 | nBo1 | nABo1 | nAe1 | nBe1 |
| … | … | … | … | … | … | … | … | … |
| ti | nAi | nBi | nTi | nAoi | nBoi | nABoi | nAei | nBei |
| … | … | … | … | … | … | … | … | … |
| tn | nAn | nBn | nTn | nAon | nBon | nABon | nAen | nBen |
| \(\sum {n_{A_o}}\) | \(\sum {nBo}\) | \(\sum {nAe}\) | \(\sum {nBe}\) |
Sob a hipótese nula de que não há diferença entre as curvas de sobrevida entre os dois estratos, o número de eventos esperados em cada estrato e em cada instante ti é igual ao produto do número total de eventos no instante ti pela proporção de indivíduos em risco em cada estrato:
\(nAe_i = nABo_i\ .\ \frac{nA_i}{nT_i}\)
\(nBe_i = nABo_i\ .\ \frac{nB_i}{nT_i}\)
A última linha da tabela mostra as somas dos valores observados (\(\sum nAo\) e \(\sum nBo\)) e esperados \((\sum nAe\) e \(\sum nBe)\) em cada estrato. A estatística para o teste log-rank é calculada pela fórmula a seguir:
\[\begin{align} \chi^2_{lr} = \frac {(\sum nAo - \sum nAe)^2} { \sum nAe } + \frac{(\sum nBo - \sum nBe)^2} {\sum nBe} \tag{20.1} \end{align}\]
A estatística (20.1) é uma variável aleatória com aproximadamente uma distribuição \(\chi^2\) com 1 grau de liberdade, quando a hipótese nula de que não há diferença entre as funções de sobrevida de cada estrato é verdadeira, desde que os nAi e nBi não sejam muito pequenos.
Valores suficientemente grandes de \(\chi_{lr}^2\) levam à rejeição de H0, ou seja, se \(\chi_{lr}^2 > \chi_{1-\alpha, 1}^2\) , então H0 é rejeitada.
Vamos ver como realizaríamos esse teste para um pequeno número de registros do conjunto de dados cancer. O resultado dos dois comandos abaixo são os 5 primeiros instantes de tempo em que ocorreram mortes entre as mulheres no conjunto de dados cancer, o número de mulheres em risco de morrer e o número de mulheres que morreram em cada instante, respectivamente.
.Survfit <- survfit(Surv(time, status, type="right") ~ 1, conf.type="log",
conf.int=0.95, type="kaplan-meier", error="greenwood",
data=cancer, subset=sex == "feminino")
head(with(.Survfit, data.frame(time, n.risk, n.event)), 5)## time n.risk n.event
## 1 5 90 1
## 2 60 89 1
## 3 61 88 1
## 4 62 87 1
## 5 79 86 1De modo análogo, o resultado dos dois comandos a seguir são os 5 primeiros instantes de tempo em que ocorreram mortes entre os homens no conjunto de dados cancer, o número de homens em risco de morrer e o número de homens que morreram em cada instante, respectivamente.
.Survfit <- survfit(Surv(time, status, type="right") ~ 1, conf.type="log",
conf.int=0.95, type="kaplan-meier", error="greenwood",
data=cancer, subset=sex == "masculino")
head(with(.Survfit, data.frame(time, n.risk, n.event)), 5)## time n.risk n.event
## 1 11 138 3
## 2 12 135 1
## 3 13 134 2
## 4 15 132 1
## 5 26 131 1A tabela 20.2 mostra como a tabela 20.1 é construída para os 5 menores tempos nos quais houve pelo menos uma morte no conjunto de dados. Os dois estratos são: A - homens, B - mulheres.
| tempo | Homens | Mulheres | Total | Homens | Mulheres | Total | Homens | Mulheres |
|---|---|---|---|---|---|---|---|---|
| 5 | 138 | 90 | 228 | 0 | 1 | 1 | 0,61 | 0,39 |
| 11 | 138 | 89 | 227 | 3 | 0 | 3 | 1,82 | 1,18 |
| 12 | 135 | 89 | 224 | 1 | 0 | 1 | 0,60 | 0,40 |
| 13 | 134 | 89 | 223 | 2 | 0 | 2 | 1,20 | 0,80 |
| 15 | 132 | 89 | 221 | 1 | 0 | 1 | 0,60 | 0,40 |
| 7 | 1 | 4,83 | 3,17 |
A primeira linha da tabela 20.2 corresponde ao tempo t1 = 5 dias. Nesse instante, 138 homens (nA1 = 138) e 90 mulheres (nB1 = 90) estavam em risco, logo 228 pessoas estavam em risco de morte. Em t1 = 5, houve uma morte entre as mulheres (nB1 = 1) e nenhuma entre os homens (nA1 = 0), logo o número total de mortes no instante 5 é igual a 1 (nAB1 = 1). O número de mortes esperadas entre os homens em t1 = 5 é dado então por nAe1 = 1 x 138/228 = 0,61. O número de mortes esperadas entre as mulheres em t1 = 5 é dado então por nBe1 = 1 x 90/228 = 0,39. De modo análogo, se obtém as outras 4 linhas da tabela, correspondentes aos tempos 11, 12, 13 e 15 dias.
O número de mortes observadas e esperadas entre os homens até o instante t = 15 dias foi de 7 \((\sum nAo = 7)\) e 4,83 \((\sum nAe = 4,83)\), respectivamente. Entre as mulheres, o número de mortes observadas e esperadas até o instante t = 15 dias foi de 1 (\(\sum nBo = 1\)) e 3,17 (\(\sum nBe = 3,17\)), respectivamente. Substituindo esses valores na expressão (20.1), obtemos:
\(\chi^2_{lr} = \frac{(7 - 4,83)^2} { 4,83 } + \frac{(1 - 3,17)^2} { 3,17 }=2,46\)
Se adotarmos o nível de significância de 5%, esse valor de \(\chi^2_{lr} < \chi_{.95, 1}^2= 3,84\), e não rejeitaríamos a hipótese nula. Nós usamos, porém, somente uma pequena porção do conjunto de dados cancer. Para fazer o teste para todos os registros do conjunto de dados cancer, usamos a seguinte opção no R Commander:
\[\text{Estatísticas} \Rightarrow \text{Análise de Sobrevida} \Rightarrow \text{Compare as funções de sobrevida}\]
Na tela Compare Funções de Sobrevida (figura 20.9), selecionamos a variável de tempo (time) e censura (status) e a variável de estratificação (sex). O valor do parâmetro rho = 0 implica que o teste será realizado como mostrado mais acima. Diferentes variantes do teste de log-rank são especificadas por outros valores para rho. Para maiores informações, vide a ajuda da função survdiff do pacote survival.
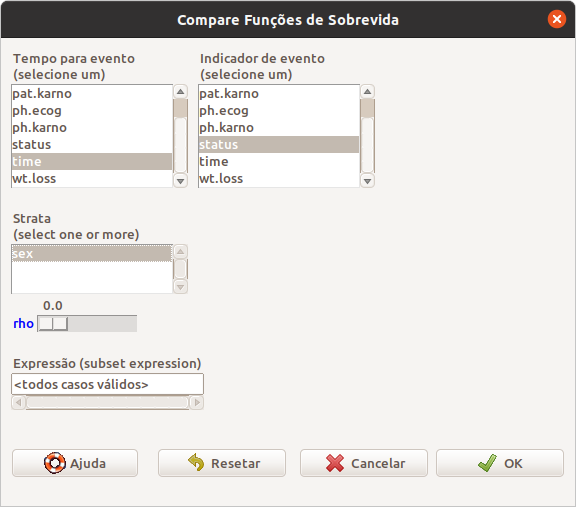
Figura 20.9: Tela para configurar as variáveis que serão utilizadas para realizar o teste log-rank para comparar as curvas de sobrevida para diferentes estratos.
A função a seguir é executada e os resultados são mostrados logo após.
survdiff(Surv(time,status) ~ sex, rho=0.0, data=cancer)## Call:
## survdiff(formula = Surv(time, status) ~ sex, data = cancer, rho = 0)
##
## N Observed Expected (O-E)^2/E (O-E)^2/V
## sex=masculino 138 112 91.6 4.55 10.3
## sex=feminino 90 53 73.4 5.68 10.3
##
## Chisq= 10.3 on 1 degrees of freedom, p= 0.001Houve 112 mortes entre os homens e 53 entre as mulheres no período de observação. O valor de \(\chi^2_{lr} = 10,3\), e o valor de p = 0,001. Se adotarmos o nível de significância de 5%, a hipótese nula de igualdade das funções de sobrevida para homens e mulheres é rejeitada.
Este capítulo é apenas uma introdução ao tema. A análise de sobrevida pode ser completada com modelos que levam em conta os efeitos de várias variáveis simultaneamente sobre a função de sobrevida. Um dos modelos mais utilizados é o modelo de riscos proporcionais de Cox. A teoria e a construção desses modelos estão fora do escopo deste texto.
O tutorial Survival Analysis in R, de Emily Zabor (Zabor 2018) inclui tópicos mais avançados da análise de sobrevida usando o R, incluindo modelos de risco proporcionais de Cox e textos de referência para aqueles que desejam se aprofundar nesse assunto.
20.8 Exercício
Com o conjunto de dados stagec do pacote rpart (GPL-2 | GPL-3) do R, faça as atividades abaixo.
- Verifique a ajuda para o conjunto de dados.
- Carregue o conjunto de dados.
- Visualize os registros do conjunto de dados.
- Obtenha a curva de sobrevida, considerando a progressão do tumor como evento de interesse.
- Obtenha as probabilidades de sobrevida em 5 e 10 anos.
- Obtenha as curvas de sobrevida para cada nível do status de ploidia do tumor. Comente os resultados.
- Compare as curvas de sobrevida em “f” pelo teste log-rank.
- Gere um relatório no R Markdown.